
表观遗传学是指在基因组的核苷酸序列不发生改变的情况下,生物个体表型却发生了可遗传性变化的现象。DNA甲基化是目前研究最深入的表观遗传修饰,简言之,是指在DNA甲基转移酶的催化下,以S-腺苷甲硫氨酸为甲基供体,将活性甲基转移至DNA链中特定碱基的化学修饰过程,在哺乳动物中,主要生成为5-甲基胞嘧啶(5-mC)。

图1-在DNA甲基化催化酶(DNMT)作用下,胞嘧啶甲基化的过程

DNA甲基化可以发生在胞嘧啶的C-5位、腺嘌呤的N-6位、鸟嘌呤的N-7位等,它们分别由不同的DNA甲基化酶催化,生成5-甲基胞嘧啶(5-mC)、N6-甲基腺嘌呤(N6-mA)及7-甲基鸟嘌呤(7-mG)。
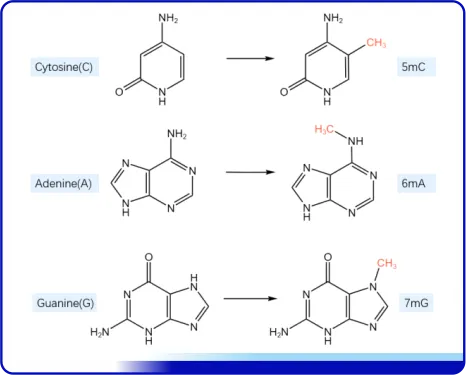
图2-碱基中甲基化发生的位置示意图

Dam甲基化:在GATC序列中腺嘌呤N6位发生甲基化。
Dcm甲基化:在CCAGG和CCTGG序列中第2个胞嘧啶C5位发生甲基化。
CpG甲基化:在真核生物(如植物和哺乳动物)中,影响酶切最常见的甲基化类型为CpG 甲基化。CpG甲基转移酶将甲基转移到胞嘧啶残基的C5位置。
当甲基化位点与限制性内切酶识别序列发生重叠,大部分酶切会受到阻断或影响。

图3-dam,dcm,CpG甲基化示意图

根据以上内切酶的甲基化敏感性,对部分甲基化敏感的限制性内切酶进行统计和分类,如下表:

备注:下划线表示内切酶识别的序列,彩色标记的碱基表示会发生甲基化修饰的序列。

随着 DNA 甲基化被发现,人们开始不断的探索如何更好更便捷地对 DNA 甲基化进行研究。对于生物体而言,基因位点的甲基化的改变可能会导致基因表达的改变,因此,对于位点上甲基化水平的研究就尤为重要。目前比较常用的就是利用甲基化敏感酶对基因组 DNA 序列进行识别切割的方法对单个位点的甲基化水平进行研究。

甲基敏感的限制性内切酶 PCR(Methyl-Sensitive Restriction Enzymes PCR):该方法较为久远,原理是甲基化敏感酶(如 HpaⅡ)对酶切位点中的 CG 甲基化敏感的特性,如果酶切位点的 CG 属于低甲基化,则可以被切开,PCR 扩增后和对照相比没有条带或者 条带很暗,相反如果酶切位点的 CG 属于高甲基化,则不会被切开,PCR 扩增后和对照 相比条带亮度相当,可以通过电泳的结果快速地判断位点甲基化水平的高低,这种方法操作简单成本低而且结果容易观察。

图4-甲基敏感的限制性内切酶检测DNA甲基化
在上述方法的基础上发展出更精确的甲基化定量分析,即使用荧光定量PCR(qPCR)对甲基化酶切后的底物进行检测。在目标位点侧翼上下游设计PCR引物,未被酶切的DNA可以作为PCR模板扩增,而被切开的DNA理论上无法扩增。剩余未酶切DNA的水平取决于目标位点的甲基化程度,位点甲基化程度越高,能被HpaII切割的片段就越少,可供PCR扩增的DNA模板就越多,在qPCR中就越早检测到DNA扩增(即Ct值越低)。根据这一原理,依据qPCR分析即可测定位点的甲基化状态。
目前比较常用的就是利用甲基化敏感酶对基因组 DNA 序列进行识别切割的方法对单个位点的甲基化水平进行研究。甲基化敏感的内切酶在甲基化分析和检测领域有着广泛的应用。下图展示了甲基化分析方法的发展历程,从液相色谱分析到二维电泳,再到3代测序技术在甲基化分析中的应用,伴随着甲基化测序技术的不断发展,甲基化敏感的内切酶参与的甲基化测序的方法也同样不断更新迭代。
目前主要应用多集中在样品的前处理步骤,通过联合亚硫酸盐测序和高通量测序等各种平台分析,从而进行整体和特定区域的甲基化水平分析。
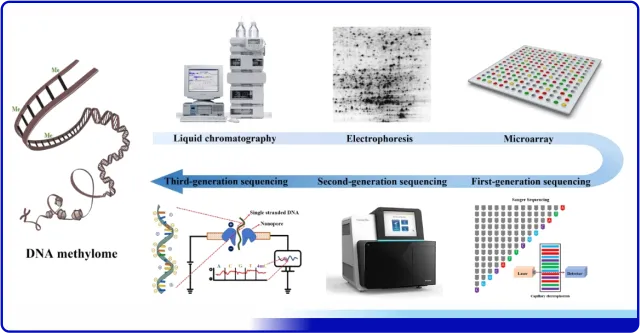
图6-甲基敏感的限制性内切酶检测DNA甲基化

产品:HinP1Ⅰ, HhaⅠ, AciⅠ, HpaⅡ, BssHⅡ, BsrFⅠ, BspEⅠ.
产品特点
特异性:酶切识别特异性强,产物清晰。
高效率:验证酶切效率高。达到或优于进口产品。
高保真:过夜酶切,星号活性极低。
宝锐甲基化敏感内切酶酶切效率高,酶切产物清晰,酶切产物的条带均一,无非特异性切割,即无星活性现象,达到或优于进口N公司产品。

参考文献:
Moore LD, Le T, Fan G. DNA methylation and its basic function. Neuropsychopharmacology. 2013 Jan;38(1):23-38.Mattei AL, Bailly N, Meissner A. DNA methylation: a historical perspective. Trends Genet. 2022 Jul;38(7):676-707.Meng H, Cao Y, Qin J, Song X, Zhang Q, Shi Y, Cao L. DNA methylation, its mediators and genome integrity. Int J Biol Sci. 2015 Apr 8;11(5):604-17.Fitz-James M H, Cavalli G. Molecular mechanisms of transgenerational epigenetic inheritance[J].Nature reviews. Genetics. 2022.Petryk N, Bultmann S, Bartke T, et al. Staying true to yourself: mechanisms of DNA methylationmaintenance in mammals[J]. Nucleic Acids Research. 2021, 49(6): 3020-3032.Antunes C, Da Silva J D, Guerra-Gomes S, et al. Tet3 ablation in adult brain neurons increasesanxiety-like behavior and regulates cognitive function in mice[J]. Molecular Psychiatry. 2021, 26(5):Singer-Sam J, LeBON JM, Tanguay RL, Riggs AD. A quantitative HpaII-PCR assay tomeasure methylation of DNA from a small number of cells. Nucleic acids research.Li S, Tollefsbol TO. DNA methylation methods: Global DNA methylation and methylomic analyses. Methods. 2021 Mar;187:28-43.